Des scientifiques du Massachusetts Institute of Technology (MIT) ont récemment publié un puissant modèle d'intelligence artificielle open source, appelé Boltz-1. Cette innovation promet d'accélérer considérablement la recherche biomédicale et le développement de médicaments.
Boltz-1 est le premier modèle entièrement open source à atteindre le même niveau de performance qu'AlphaFold3 de Google DeepMind dans la prédiction de structures biomoléculaires. L'équipe de développement, basée à la clinique Jameel d'apprentissage automatique pour la santé du MIT, est dirigée par les étudiants diplômés Jeremy Walrond et Gabriel Corso, en collaboration avec le chercheur du MIT, Salo Pasaro, et les professeurs de génie électrique et informatique Regina Barzilay et Tommy Jaakkola.
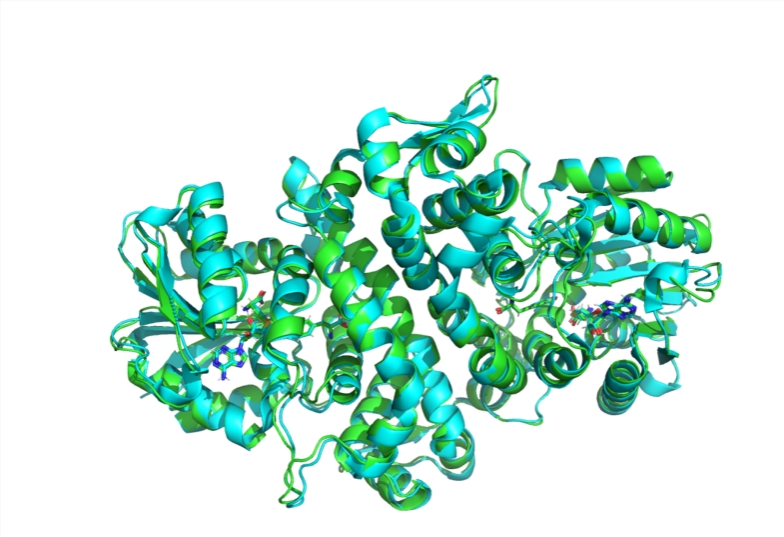
Lors d'une présentation le 5 décembre, Walrond et Corso ont déclaré que leur objectif ultime était de favoriser la collaboration mondiale, d'accélérer les découvertes scientifiques et de fournir une plateforme robuste pour faire progresser la modélisation biomoléculaire. Corso a mentionné : « Nous espérons que cela servira de point de départ pour la communauté », soulignant que le nom « Boltz-1 » plutôt que « Boltz » vise à encourager la participation de la communauté.
Les protéines jouent un rôle crucial dans presque tous les processus biologiques, et leur forme est étroitement liée à leur fonction. Comprendre la structure des protéines est donc essentiel pour concevoir de nouveaux médicaments ou créer de nouvelles protéines ayant des fonctions spécifiques. La prédiction précise de leur structure a longtemps représenté un défi majeur pour la communauté scientifique, en raison de la complexité extrême du processus de pliage des longues chaînes d'acides aminés des protéines en structures tridimensionnelles.
AlphaFold2 de DeepMind, grâce à l'apprentissage automatique, prédit rapidement les structures 3D des protéines avec une précision telle que les scientifiques expérimentaux ont du mal à la distinguer de la réalité. AlphaFold3, une amélioration d'AlphaFold2, utilise un modèle d'IA génératif, mais son manque d'accès open source a suscité des critiques de la part de la communauté scientifique. C'est pourquoi l'équipe du MIT a entrepris de développer Boltz-1, en suivant les principes d'AlphaFold3 tout en améliorant la précision et l'efficacité prédictive du modèle.
L'équipe de recherche a travaillé pendant quatre mois, mené de nombreuses expériences et surmonté les problèmes d'ambiguïté et d'hétérogénéité rencontrés dans les banques de données de protéines. Finalement, leurs expériences ont montré que Boltz-1 atteint la même précision qu'AlphaFold3 dans la prédiction de structures biomoléculaires complexes.
Les chercheurs prévoient d'améliorer encore les performances de Boltz-1 et de réduire les temps de prédiction. Ils invitent également les chercheurs à tester Boltz-1 sur GitHub et à échanger avec d'autres utilisateurs via un canal Slack. L'équipe espère que Boltz-1 favorisera une collaboration plus large et stimulera des applications créatives au sein de la communauté.
Projet : https://jclinic.mit.edu/democratizing-science-boltz-1/
Points clés :
🌍 Boltz-1 est le premier modèle de prédiction de structures biomoléculaires entièrement open source, atteignant les mêmes performances qu'AlphaFold3.
🧬 Le développement de ce modèle vise à favoriser la collaboration mondiale et à promouvoir la recherche biomédicale et le développement de médicaments.
🔬 L'équipe du MIT espère que Boltz-1 simplifiera la prédiction des structures protéiques, permettant à un plus grand nombre de chercheurs d'utiliser cet outil puissant.
