मैसाचुसेट्स इंस्टीट्यूट ऑफ टेक्नोलॉजी (MIT) के वैज्ञानिकों ने हाल ही में एक शक्तिशाली ओपन-सोर्स आर्टिफिशियल इंटेलिजेंस मॉडल लॉन्च किया है, जिसका नाम Boltz-1 है। यह नवाचार जैव चिकित्सा अनुसंधान और दवा विकास को महत्वपूर्ण रूप से तेज करने की उम्मीद है।
Boltz-1 पहला पूरी तरह से ओपन-सोर्स मॉडल है, जो जैविक अणुओं की संरचना की भविष्यवाणी में गूगल की DeepMind के AlphaFold3 के समान उन्नत स्तर तक पहुंच सकता है। इस मॉडल के विकास टीम MIT Jameel मशीन लर्निंग स्वास्थ्य क्लिनिक से है, जिसमें मुख्य रूप से शोध छात्र जेरमी वॉलवेंड और गैब्रिएल कोर्सो शामिल हैं, और सहयोगी टीम में MIT के शोधकर्ता सारो पासारो और इलेक्ट्रिकल इंजीनियरिंग और कंप्यूटर साइंस के प्रोफेसर रेगिना बेजली और टॉमी याकारा भी शामिल हैं।
5 दिसंबर को एक प्रेस कॉन्फ्रेंस में, वॉलवेंड और कोर्सो ने कहा कि उनका अंतिम लक्ष्य वैश्विक सहयोग को बढ़ावा देना, वैज्ञानिक खोजों को तेज करना, और जैविक अणु मॉडलिंग को आगे बढ़ाने के लिए एक मजबूत मंच प्रदान करना है। कोर्सो ने कहा, "हम आशा करते हैं कि यह समुदाय के लिए एक प्रारंभिक बिंदु बनेगा," और इस बात पर जोर दिया कि "Boltz-1" नामकरण का उद्देश्य समुदाय की भागीदारी को प्रोत्साहित करना है।
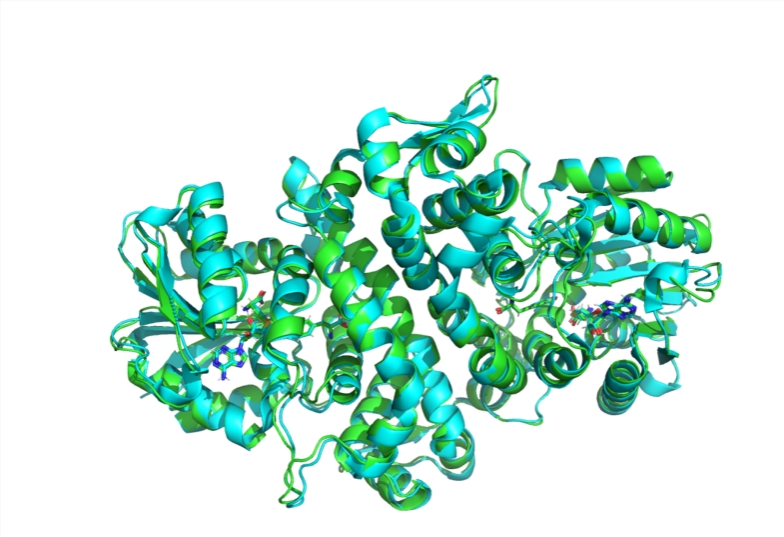
प्रोटीन लगभग सभी जैविक प्रक्रियाओं में महत्वपूर्ण भूमिका निभाते हैं, और प्रोटीन का आकार इसके कार्य से निकटता से संबंधित है, इसलिए नए दवाओं के डिजाइन या विशेष कार्यों वाले नए प्रोटीन की इंजीनियरिंग के लिए प्रोटीन की संरचना को समझना अत्यंत महत्वपूर्ण है। प्रोटीन के लंबे श्रृंखला अमीनो एसिड का तीन-आयामी संरचना में मुड़ने की प्रक्रिया बेहद जटिल है, और इसकी संरचना की सटीक भविष्यवाणी करना वैज्ञानिक समुदाय के सामने एक बड़ा चुनौती रहा है।
DeepMind का AlphaFold2 मशीन लर्निंग का उपयोग करके 3D प्रोटीन संरचना की तेजी से भविष्यवाणी करता है, जिसकी सटीकता इतनी अधिक है कि प्रयोगात्मक वैज्ञानिकों के लिए इसे पहचानना मुश्किल है। AlphaFold3 ने इस आधार पर सुधार किया है, जो जनरेटिव AI मॉडल का उपयोग करता है, लेकिन चूंकि यह पूरी तरह से ओपन-सोर्स नहीं है, इसलिए इसे वैज्ञानिक समुदाय द्वारा आलोचना का सामना करना पड़ा। इसलिए, MIT की शोध टीम ने Boltz-1 विकसित करने का निर्णय लिया, AlphaFold3 के मूल विचारों का पालन करते हुए और इसके आधार पर सुधार करते हुए मॉडल की सटीकता और भविष्यवाणी की दक्षता को बढ़ाया।
शोध टीम ने चार महीने का समय बिताया, कई प्रयोग किए, और प्रोटीन डेटा बैंक में अस्पष्टता और विषमता की समस्याओं को पार किया। अंततः, उनके प्रयोगों ने दिखाया कि Boltz-1 जटिल जैविक अणु संरचना की भविष्यवाणी में AlphaFold3 के समान सटीकता तक पहुंच गया है।
शोधकर्ता Boltz-1 के प्रदर्शन को बढ़ाने और भविष्यवाणी के समय को कम करने की योजना बना रहे हैं। उन्होंने शोधकर्ताओं को GitHub पर Boltz-1 का परीक्षण करने और अन्य उपयोगकर्ताओं के साथ बातचीत करने के लिए Slack चैनल के माध्यम से आमंत्रित किया है। शोध टीम आशा करती है कि Boltz-1 व्यापक सहयोग को बढ़ावा देगा और समुदाय की रचनात्मक अनुप्रयोगों को प्रेरित करेगा।
प्रोजेक्ट: https://jclinic.mit.edu/democratizing-science-boltz-1/
मुख्य बातें:
🌍 Boltz-1 पहला पूरी तरह से ओपन-सोर्स जैविक अणु संरचना भविष्यवाणी मॉडल है, जो AlphaFold3 के समान प्रदर्शन तक पहुंचता है।
🧬 इस मॉडल का विकास वैश्विक सहयोग को बढ़ावा देने और जैव चिकित्सा अनुसंधान और दवा विकास को आगे बढ़ाने के लिए किया गया है।
🔬 MIT टीम Boltz-1 के माध्यम से प्रोटीन संरचना की भविष्यवाणी को सरल बनाना चाहती है, ताकि अधिक शोधकर्ता इस शक्तिशाली उपकरण का उपयोग कर सकें।



